Fallstudie: Ein Model für biomechanische Simulations-Daten
Diese Fallstudie beschreibt meine Beiträge im Kontext der relevanten Projekt-Ergebnisse. Viele meiner Arbeiten fanden während meiner Zeit bei NEC statt; danach bearbeitete ich freiberuflich Unteraufträge des Projekts.
Simulations-Daten und ihre Beziehungen
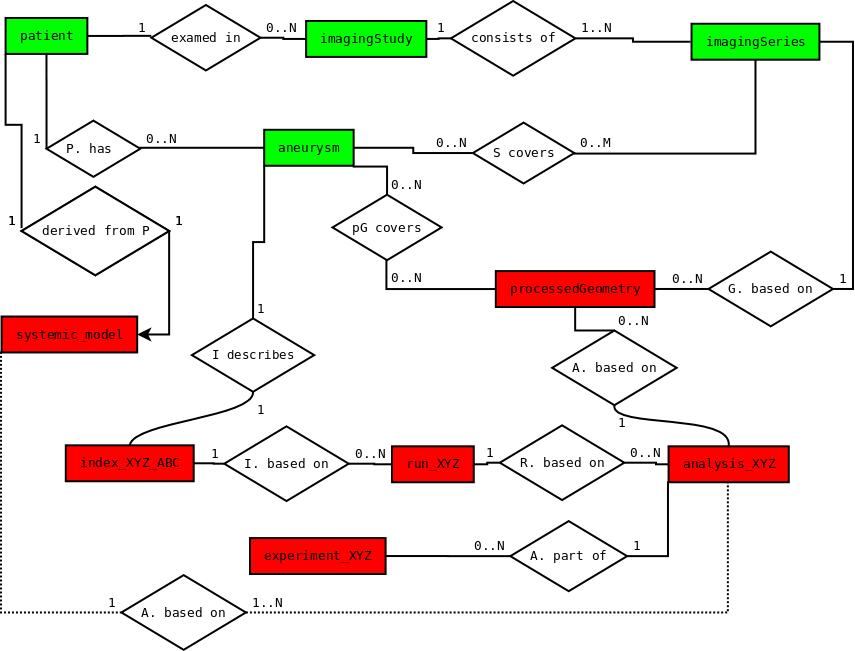
Die biomechanische Werkzeugkette von @neurIST erzeugt eine Menge Daten: Temporäre und endgültige, strukturierte und unstrukturierte, riesige und winzige, teuer oder billig zu erzeugende. Auf der Input-Seite stehen klinische Daten: medizinische Bilddaten, aber auch andere gemessene physiologische Größen wie Blutdruck oder Hämatokrit-Werte. Die wichtigsten Klassen von Daten sind medizinische Bilder, Aneurysmen (als die hier interessierende anatomische Struktur), geometrische Repräsentationen von Aneurysmen (diese sind in den medizinischen Bilder nur implizit repräsentiert und müssen mühsam herausgearbeitet werden), biomechanische Analysen (Simulationen, siehe unten), und potentielle Risiko-Faktoren, d.h. biomechanische Charakterisierungen von Aneurysmen.
Die Beziehungen zwischen diesen Datenklassen ist nicht hierarchisch, insbesondere da wir auch Varianten zulassen: Es kann mehrere Aneurysmen pro Patient geben, und verschiedene medizinische Bilder des Patienten zu verschiedenen Zeitpunkten, die nicht notwendigerweise jeweils alle Aneurysmen abbilden. Aus den Bildern werden geometrische Darstellungen der interessierenden Blutgefäße erzeugt, die u. U. wiederum nur einen Teil der Aneurysmen enthalten. Darauf aufbauend werden biomechanische Simulationen spezifiziert, zum Beispiel eine Blutflussanalyse. Eine solche bedingt eine entsprechendes Abschneiden der Geometrie, Abgrenzen des eigentlichen Aneurysmas vom Rest (indem eine Art Hals oder Trennfläche definiert wird), Definition von Randbedingungen und weitere Festlegungen (siehe auch die Fallstudie zur Modellierung).
Datenmodellierung
Das Kernproblem der Datenmodellierung ist es, die richtigen Datenklassen zu finden: nicht zu grob und nicht zu feinkörnig. Die folgenden Fragen helfen dabei:
- Welche Aspekte könnten unabhängig voneinander geändert oder benutzt werden?
- Kann es 1:n oder sogar m:n Beziehungen zwischen Klassen von Daten geben?
Ein gutes Beispiel ist die Darstellung verschiedener Teilmengen einer Geometrie, die (evtl. mehrere) Aneurysmen und die umgebendend Blutgefäße darstellt. Für verschiedene Analysen werden unterschiedlich große Teilmengen benötigt, die durch Abschneiden einer Maximal-Geometrie erzeugt werden. Wir könnten jetzt diese Teilgeometrien als vorrangige Datenklassen behandeln und abspeichern. Stattdessen speichern wir aber nur einmal die Maximal-Geometrie und dann für jede Teilmenge einfach die Spezifikation für das Abschneiden (woraus Teilgeometrien einfach generiert werden können). Damit werden diese Spezifikationen "Daten erster Klasse" und können einfach kopiert oder manipuliert werden, z.B. um die Geometrien etwas zu vergrößern oder zu verkleinern.
Ein anderes Beispiel von weitreichender Bedeutung ist die Spezifikation einer biomechanischen Simulation. Es ist vorteilhaft, die Spezifikation des Simulations-Problems (analysis) von seiner Ausführung (run) zu trennen. Denn für eine einzige Spezifikation kann es viele verschieden Läufe geben, die verschiedene Anwendungen oder numerische Parameter benutzen (das kann man zur Konvergenzanalyse oder Verifikation benutzen). Deswegen wurden die Spezifikation in einem sog. abstract problem description (APD) getrennt von den eigentlichen Läufen modelliert. Diese Beschreibung ist abstrakt im dem Sinne, dass sie unabhängig von den konkreten Simulationspaketen ist, mit denen eine Lösung berechnet werden kann. Sie ist aber vollständig, sodass für jedes geeignete Paket der nötige Input prinzipiell automatisch generiert werden kann (siehe die Beschreibung der Automatisierung der Werkzeugkette).
Eine wichtige Klasse zum Gruppieren von Analysen oder Problem-Spezifikationen ist experiment. Oft ist es wünschenswert, verschiedene Varianten von Analysen zu berechnen (z.B. um den Effekt verschiedener Modellierungsarten vergleichen). Für eine statistischen Analyse müssen diese Varianten jedoch strikt getrennt werden, um nicht "Äpfel mit Birnen zu vergleichen".
Die Beziehungen der wichtigsten Klassen werden in einem Entity Relationship Diagramm dargestellt. Die detaillierte Ausarbeitung führt zu einer SQL-basierten Datendefinition. Das Schema wurde benutzt, um die Resultate und Input-Daten von mehreren Hundert Fällen zu verwalten (als DBMS wurde MySQL benutzt). Diese Datenbank verknüpft die Simulationsdaten einerseits mit den zugrundeliegenden klinischen Daten und andererseits mit statistischen Analyse-Tools.
Ein Blick nach vorne
Die Details dieser Arbeit sind sicher spezifisch für die biomechanische Simulation von Aneurysmen. Die Konzept tragen aber weiter und können einfach auf großskalige Simulations-Systeme übertragen werden. Die Crux ist es, die richtigen Abstraktionen zu finden, damit die automatische Manipulation und Transformation von Beschreibung möglich wird, z. B. um einfach Varianten von Analysen zu generieren. Eine zentrale derartige Abstraktion ist hier die anwendungs-unabhängige Spezifikation von biomechanischen Simulationen. Das führt zu einem äußerst mächtigen System, das man als eine Art virtuelle Datenbank von Analyse-Resultaten betrachten kann: Bereits berechnete Resultate, oder die unendliche Vielfalt derer, die man durch Variation der Spezifikationen erzeugen kann.
Zum Beispiel wäre es nun möglich zu fragen, ob gewisse blutverdünnende Medikamente einen Einfluss auf das Ruptur-Risiko eines Aneurysmas haben: Eine Teilpopulation der Fälle in der Datenbank wird ausgewählt, die APDs werden so transformiert, dass die Blutviskosität entsprechend der Wirkung des Medikaments herabgesetzt wird, und die Ergebnis-Risiko-Faktoren werden verglichen. Das ist ein typisches Beispiel für "multi-case analysis": Ausgehend von den zuvor erstellten Artefakten wie Geometriebeschreibung und Problem-Spezifikation können Varianten automatisch generiert und berechnet werden, wobei der manuelle Aufwand unabhängig von der Anzahl der Fälle ist.
Meine Beiträge
Für die Daten der @neurIST-Werkzeugkette habe ich ein semantisches Modell entwickelt. Die involvierten Prozesse und Datenklassen habe ich spezifiziert und jeden Prozess mit einer präzisen Definition seiner Ein- und Ausgabe-Daten versehen. Zusammen mit Christian Ebeling (FhG SCAI) habe ich das Datenmodell mit Hilfe von SQL formalisiert. Das resultierende Datenbank-Schema umfasst etwa 80 Tabellen und enthält seine eigene Dokumentation in Meta-Tabellen. Klinische Daten (die vom Projekt in einem anderen Schema beschrieben wurden) können damit eindeutig den entsprechenden Simulationsdaten zugeordnet werden. Die Arbeit verlangte intensive Zusammenarbeit mit Projektpartnern, die für folgende Bereiche zuständig waren:
- Die verschiedene Details der Werkzeugkette
- Die Verwaltung der klinischen Daten, inklusive der rechtlichen Einschränkungen
- Die statistische Auswertung der Daten zum Auffinden von Risiko-Faktoren
Für meine Entwicklung hatte ich mir von vornherein das Ziel gesetzt, ein multi-case Szenario zu unterstützen: Für eine große Anzahl Fälle sollte es möglich sein, die abstrakten Problem-Beschreibungen (APDs) automatisch zu manipulieren und in Varianten zu transformieren (z.B. indem Randbedingungen ersetzt werden). Diese neuen Problem-Beschreibungen können dann automatisch ausgeführt und ausgewertet werden. Ein solches System ist ein äußerst mächtiges Werkzeug für Forscher und Ingenieure, die neue simulations-gestützte Experimente auf der Datenbasis durchführen wollen: Die notwendige manuelle Arbeit wird signifikant reduziert und ist im Idealfall unabhängig von der Anzahl der Fälle.
Viele wissenschaftliche Untersuchungen werden erst mit einem solchen System praktikabel!